นักวิทยาศาสตร์จากสถาบันชีวคำนวณแห่งมหาวิทยาลัยซานฟรานซิสโก ร่วมมือกับห้องปฏิบัติการแสงซินโครตรอนแห่งเบิร์คเลย์และสแตนฟอร์ด สหรัฐอเมริกาค้นพบตัวยับยั้งที่เป็นกุญแจสำคัญในการจับคู่กับตำแหน่งของโปรตีน macro domain ซึ่งมีบทบาทสำคัญในการจำลองตัวเองและการเพิ่มจำนวนภายในร่างกายของมนุษย์ของไวรัสโคโรนาก่อโรคโควิด-19
Assoc.Prof. James Fraser หัวหน้าโครงการวิจัย เปิดเผยว่าการทำงานของโปรตีน macro domain ของเชื้อไวรัสก่อโรคโควิด-19 ยังไม่เป็นที่เข้าใจอย่างชัดเจน เช่นเดียวกับโปรตีนตัวอื่นของไวรัส อาทิ โปรตีเอส (protease) หรือโปรตีนสไปก์ (spike protein) ที่กำลังอยู่ในระหว่างการศึกษาอยู่เช่นกัน การหาตัวเลือกที่เหมาะสมในการสร้างยาที่สามารถยับยั้งความสามารถของไวรัสในการจำลองตัวเองและเพิ่มจำนวนในร่างกายมนุษย์จึงมีความสำคัญมาก
ขณะนี้ทางทีมวิจัยใช้เทคนิค X-Chem screening ในการค้นหาโมเลกุลขนาดเล็กที่มีลักษณะคล้ายคลึงกับกุญแจตัวยับยั้งเพื่อเป็นต้นแบบในการพัฒนายาต้านไวรัส โดยในขณะนี้ทีมวิจัยกำลังเตรียมตีพิมพ์รายงานลงวารสารวิชาการ และได้เผยแพร่ โครงสร้างภาพสามมิติของโปรตีน macro domain ของเชื้อไวรัสก่อโรคโควิด-19 ทางออนไลน์ เพื่อเป็นข้อมูลต้นแบบในการพัฒนายาต้านเชื้อไวรัสโคโรนา ซึ่งทางสถาบันแสงซินโครตรอนไดมอนด์แห่งประเทศอังกฤษ (Diamond Light Source) ได้อาศัยโครงสร้างสามมิตินี้ในการหาโมเลกุลขนาดเล็กที่สามารถหยุดยั้งการทำงาน (ข้อมูลเลี้ยวเบนของโครงสร้างโปรตีนจับคู่กับโมเลกุลขนาดเล็ก)
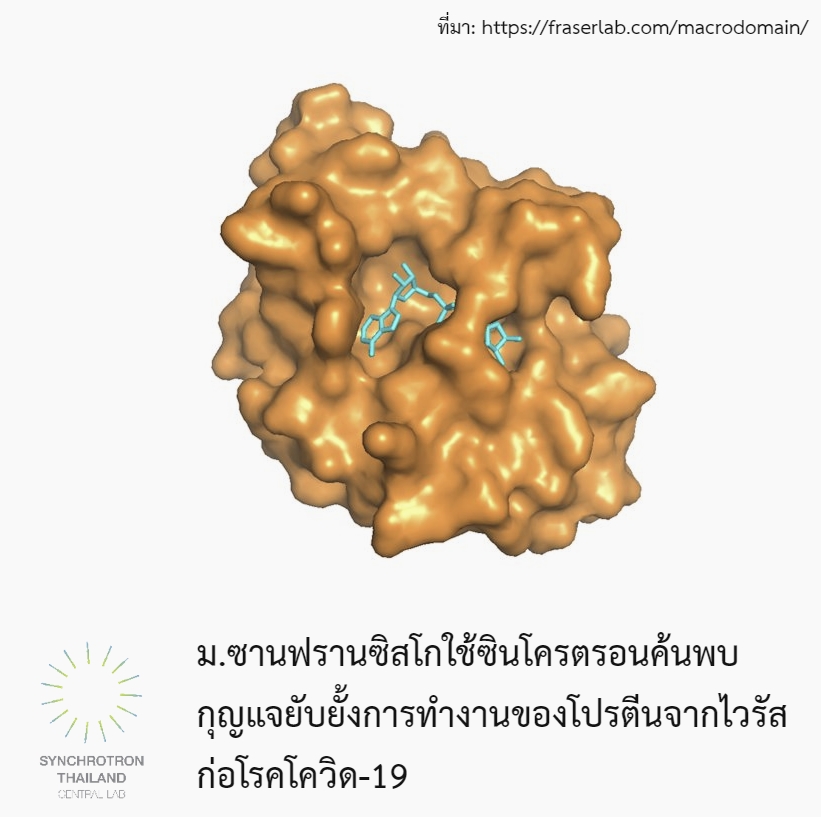
ในขณะเดียวกันทีมวิจัยของ Prof. Alan Ashworth ผู้อำนวยการของศูนย์วิจัยมะเร็งแห่งมหาวิทยาลัยซานฟรานซิสโก ได้กำลังศึกษาโครงสร้างทางชีววิทยาของเชื้อไวรัสโคโรนา และเปิดเผยว่าเอนไซม์ PARPs (Poly ADP-ribose polymerase) มีบทบาทสำคัญในการซ่อมแซมดีเอ็นเอของมนุษย์และเป็นเป้าหมายของการพัฒนายาในการยับยั้งมะเร็ง เนื่องจากเอนไซม์ชนิดนี้ทำหน้าที่ติดฉลากโมเลกุล ADP-ribose ให้กับโปรตีนภายในเซลล์มะเร็งหรือไวรัส ทำให้ระบบภูมิคุ้มกันของร่างกายสามารถเข้าทำลายสิ่งแปลกปลอมได้ หากแต่เชื้อไวรัสก่อโรคโควิด-19 มีโปรตีน macro domain ที่ถูกวิวัฒนาการให้เพิ่มความสามารถในการถอนฉลาก ADP-ribose ได้ด้วยตัวเอง ทีมวิจัยจึงพัฒนายาเพื่อหยุดการถอนฉลาก ADP-ribose เพื่อให้ร่างกายสามารถกำจัดเชื้อไวรัสโคโรนาได้ ทีมวิจัยของ Fraser ศึกษาโครงสร้างโปรตีน macro domain เพื่อค้นหาโมเลกุลขนาดเล็กที่มีรูปทรงพอดีที่จะจับคู่กับบริเวณเกิดปฏิกิริยาของโปรตีนตัวนั้น เป็นขั้นตอนแรกในการพัฒนายาต้านไวรัสซึ่งสามารถยับยั้งการเพิ่มจำนวนของไวรัสด้วยการกระตุ้นการทำงานของเอนไซม์ PARPs
การหาโครงสร้างผลึกของโปรตีนด้วยรังสีเอกซ์ (X-ray crystallography) เป็นเทคนิคสำคัญที่นำไปสู่การค้นพบนี้ โดยนักวิจัยจะตกผลึกโปรตีน macro domain แล้วนำไปวิเคราะห์โดยใช้รังสีเอกซ์ที่ห้องปฏิบัติการของสถาบันแสงซินโครตรอนในเบิร์คเลย์ (ALS) และสถาบันแสงซินโครตรอนแห่งสแตนฟอร์ด (SLAC) เพื่อเก็บข้อมูลภาพการเลี้ยวเบนของรังสีเอกซ์ที่มีลักษณะเฉพาะตัวของโปรตีนนั้น จากนั้นนำไปประมวลผลด้วยคอมพิวเตอร์ออกมาเป็นภาพโครงสร้างสามมิติของโปรตีน macro domain ในระดับอะตอมขนาด 1 อังสตรอม (เทียบเท่ากับหนึ่งในล้านของเส้นผ่าศูนย์กลางของเส้นผมมนุษย์)
ยิ่งไปกว่านั้น นักวิจัยยังใช้คอมพิวเตอร์สมรรถนะสูงในการค้นหาโมเลกุลจากคลังข้อมูลโมเลกุลขนาดเล็กทั่วโลกแล้วนำมาทดสอบจับคู่กับโครงสร้างสามมิติของโปรตีน macro domain ที่สร้างขึ้นและค้นพบโมเลกุลขนาดเล็กจำนวน 13 หน่วยที่สามารถเข้าจับกับโปรตีน macro domain ได้ โดยในกลุ่มนี้มีโมเลกุลขนาดเล็กถึง 4 หน่วยที่เข้าจับคู่กับตำแหน่งการเกิดปฏิกิริยาของโปรตีนนี้ได้อย่างพอดี ทำให้โมเลกุลเล็กกลุ่มนี้มีความเป็นไปได้สูงในการนำพัฒนายาต้านไวรัสก่อโรคโควิด-19 ต่อไป
ผู้เรียบเรียง : ดร.จักรี์รดา อัตตรัถยา

